Alterações cromossômicas
Alterações cromossômicas
Publicado em 2 de agosto de 2023
As alterações genéticas são frequentemente associadas com doenças ou síndromes. Existem diversos tipos dessas alterações, sendo a quantidade de material genômico envolvido um dos fatores por trás de seu impacto clínico. Dentre eles, os dois tipos principais são “variantes de nucleotídeo único” e “alterações cromossômicas”.
As “variantes de nucleotídeo único” são caracterizadas por afetarem poucos pares de bases (ou “letras”) do código do DNA, de forma que a sua associação com doenças depende, principalmente, da localização no genoma. Já as alterações cromossômicas geralmente atingem milhões de pares de bases de DNA. Quando uma grande quantidade de material genético é alterada, aumenta-se o risco de ocorrência de síndromes.
Como são classificadas as alterações cromossômicas?
Essas alterações podem ser classificadas como numéricas ou estruturais. Quando o genoma do indivíduo apresenta uma quantidade de cromossomos inteiros em excesso ou faltando, chamamos essa variação de “numérica”. O exemplo mais conhecido de alteração cromossômica numérica é a síndrome de Down, que é causada pela trissomia do cromossomo 21, ou seja, um cromossomo 21 a mais no genoma.
Por outro lado, as alterações cromossômicas estruturais acontecem quando pedaços (ou “segmentos”) de cromossomos estão a mais ou a menos, ou ainda, quando eles mudam de lugar. As alterações cromossômicas estruturais mais frequentes implicam em duplicações ou deleções de grandes segmentos genômicos, que geralmente envolvem diversos genes. Para que essas alterações ocorram, acontecem quebras no segmento de DNA que compõe o cromossomo. Qualquer cromossomo pode sofrê-las, havendo grande heterogeneidade de tamanhos de segmentos genômicos afetados e, consequentemente, de alterações clínicas associadas.
Tipos de avaliação de alterações cromossômicas
Os exames de análises cromossômicas geralmente são solicitados quando o indivíduo possui uma malformação congênita associada a alterações no neurodesenvolvimento, sem que haja suspeita de uma síndrome genética causada por variantes de nucleotídeo único. Outros casos em que a avaliação de alterações cromossômicas pode auxiliar no diagnóstico são atraso da puberdade, baixa estatura, infertilidade ou abortos de repetição.
O estudo dos cromossomos surgiu com a citogenética clássica, que se utiliza do exame do cariótipo por bandamento G. Apesar de ser comumente utilizado como primeira triagem diagnóstica para alterações cromossômicas, o exame apresenta uma baixa resolução. Por isso, ele impede a determinação precisa da localização de quebras cromossômicas ou o tamanho do segmento genômico afetado pelas alterações estruturais.
Nos últimos anos, o estudo de alterações cromossômicas estruturais com metodologias moleculares de alta resolução tem permitido o reconhecimento de genes e de regiões genômicas responsáveis por determinadas características humanas. Em muitos casos, após a identificação do segmento cromossômico duplicado ou deletado, é possível oferecer um melhor prognóstico e aconselhamento genético às pessoas com alterações cromossômicas estruturais e suas famílias.
Pelo fato de as alterações estruturais serem mais complexas e heterogêneas, elas são estudadas com maior frequência na pesquisa científica. Esses estudos se propõem a correlacionar o segmento cromossômico afetado pela alteração estrutural com o quadro clínico do indivíduo, o que é chamado de “correlação genótipo-fenótipo”. Para que essa correlação aconteça, os pontos de quebra dos cromossomos precisam ser definidos com alta resolução por metodologias moleculares.
Testes de alta resolução para avaliação de pontos de quebra cromossômica
Para melhor delineamento molecular e identificação das alterações cromossômicas, diversas técnicas foram desenvolvidas e aprimoradas. A principal delas é o array genômico, uma técnica de alta resolução que analisa o genoma em sua totalidade, incluindo detecção de ganhos e perdas no material genético.
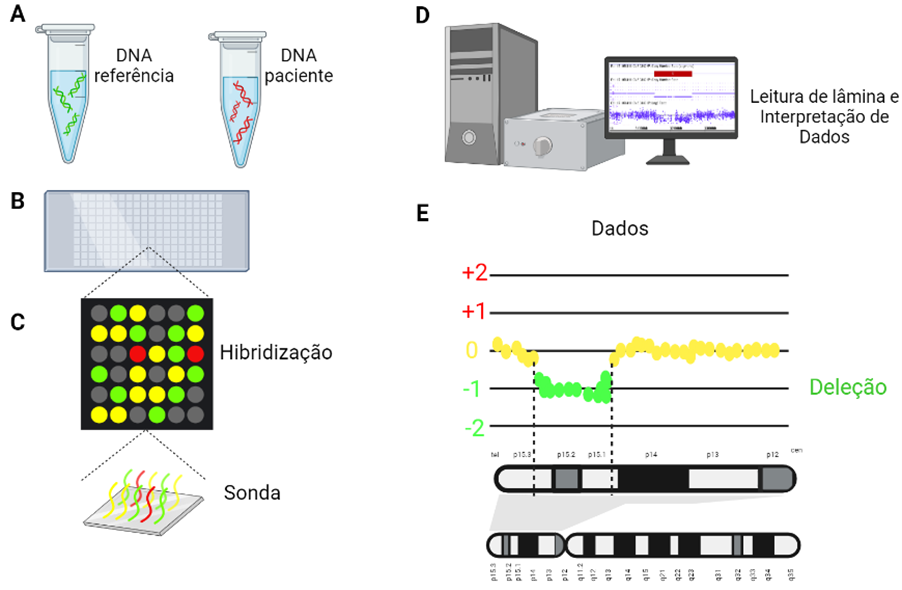
Existem alguns tipos de array genômico, como por exemplo o array CGH (do inglês, Comparative Genomic Array), demonstrado na imagem abaixo.
O procedimento do array CGH requer duas amostras de DNA, uma amostra de referência (ou seja, material genético de uma pessoa sem alterações cromossômicas) e uma amostra do paciente. Essas amostras de DNA são geralmente obtidas a partir de uma coleta de sangue. Os fragmentos genômicos são marcados com fluorescência, neste caso, verde para DNA referência e vermelho para a amostra do paciente.
Em uma lâmina, há a disposição de milhares de segmentos pequenos de DNA, contendo sequências complementares a regiões do genoma, chamadas de “sondas”. Essas sondas servem para avaliar a quantidade de material genético dos seus alvos genômicos, determinando se há perdas ou ganhos de DNA no genoma do paciente.
Como demonstrado na imagem abaixo, nas regiões em que o número de cópias no DNA da amostra teste (do paciente) for igual ao número de cópias da amostra de referência, o verde e vermelho são misturados de forma equivalente, e as sondas ficarão amarelas. Já nas regiões em que houver uma deleção no paciente, as sondas ficarão verdes, pois a fluorescência do DNA de referência vai se sobressair. Se houver duplicação no paciente, as sondas ficarão vermelhas.
A avaliação da emissão de fluorescência ocorre depois que os segmentos de DNA provenientes das amostras de material genético se ligarem às sondas. A lâmina de array é então analisada por um software, que quantifica a fluorescência emitida por cada sonda e a traduz em número de cópias de DNA do genoma do paciente.